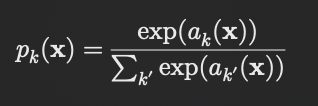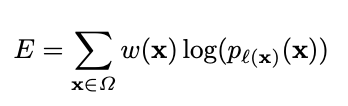U-Net:Convolutional Networks for Biomedical Image Segmentation
<논문리뷰> U-Net:Convolutional Networks for Biomedical Image Segmentation
Summary
- a network and training strategy that relies on the strong use of data augmentation to use the available annotated samples
- consists of a contracting path (encoder) to capture context and a symmetric expanding path (decoder) that enables precise localization
- can be trained end-to-end from very few images and fast
Introduction
CNN의 training set size, network size 제한으로 인해 Krizhevsky는 8개 layers, 수백만개 parameters로 이루어진 large network를 ImageNet dataset의 training image로 supervised training을 진행해 더 크고 깊은 네트워크를 훈련했다.
⇒ CNN의 classification한 이미지의 결과는 single class label이다. 하지만 Biomedical Image Segmentation와 같은 많은 시각작업에서는 ‘localization’이 포함되어야 해서 each pixel에 class label이 할당되어야 한다. biomedical domain에서는 수천장의 train imageset를 확보하는게 어렵기 때문에 Ciresan는 픽셀 주위의 local patch를 input으로 제공해, 각 픽셀의 class label을 예측하는 sliding-window setting에서 네트워크를 훈련시키는 방법을 고안했다.
- sliding window
- Benefits
- localize이 가능하고, patches로 인해 training data가 training image의 수보다 훨씬 커진다.
- Drawbacks
- 각 patch 별로 네트워크가 실행되어야하기 때문에 매우 느리고, overlapping patches 때문에 많은 중복 발생한다. 또한, 큰 size의 patches는 localization accuracy를 낮추는 max-pooling 레이어가 더 많이 필요해 localization의 정확도가 떨어지고, 작은 size의 patches는 네트워크가 볼 수 있는 context가 적어 localization 정확도와 context의 사용 사이에 trade-off가 발생한다.
In this paper?
: Fully Convolutional Network 구조를 기반으로, 매우 적은 training images로 동작하고, 더 정확한 segmentation을 생성하는 네트워크를 구축했다.
fully convolutional network의 Main Idea
polling operator가 upsampling operator로 대체되는 일반적인 contracting 네트워크를 연속적인 계층으로 보완해 출력의 해상도를 높인다. localize하기 위해, contracting path의 high resolution features는 upsampled 출력과 결합된다.(Skip Architecture). 그 다음, 연속적 컨볼루션 레이어는 이 정보를 기반으로 더 정확한 출력을 모은다.
U-Net Architecture의 important modification
‘upsampling’ 파트에서 네트워크가 context 정보를 higher resolution layers로 ‘’전파’할 수 있는 많은 수의 feature 채널을 포함한다. 그 결과, 확장경로가 수축경로와 대칭적이게 돼서 u-모양 구조가 형성된다. 네트워크는 어떠한 완전연결레이어를 포함하지 않으며, padding 없이 convolution을 적용하여 valid part만 사용한다.
즉, segmentation map은 오직 input 이미지에서 full context가 가능한 픽셀만을 포함한다.
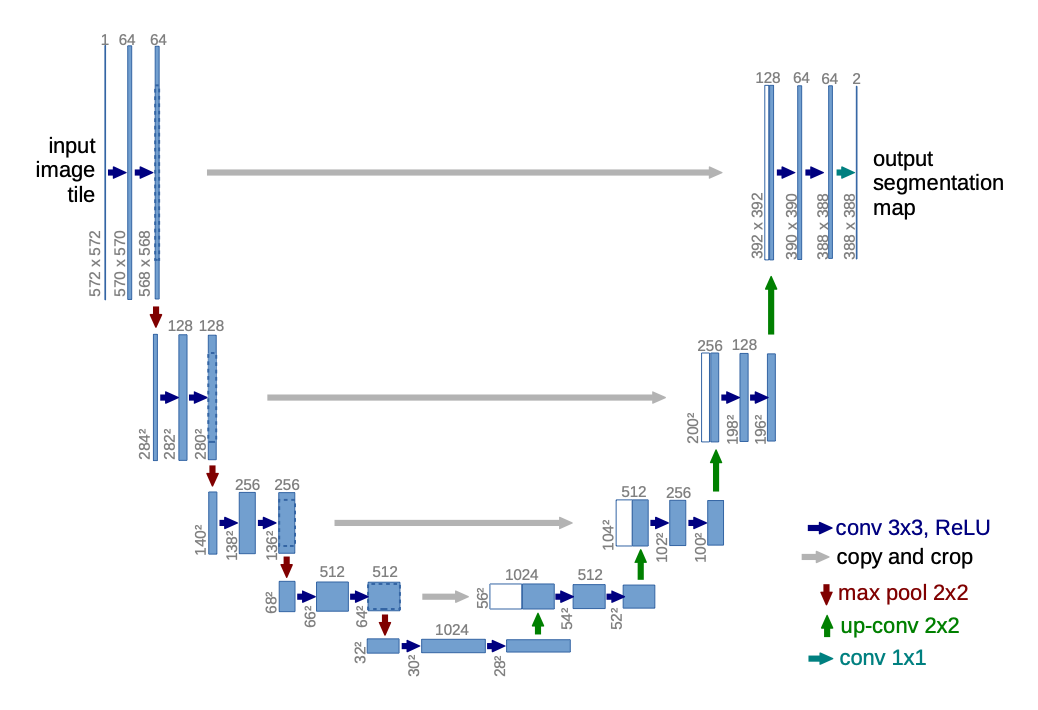
Network Architecture
구성요소
‘왼쪽의 contracting path + 오른쪽의 expansive path’로 구성된다.
전형적인 convolutional network의 아키텍처(without fully connected layer)
- Downsampling
- 3×3 Convolution Layer + ReLu + Batch Normalization (No Padding, Stride 1) x 2
⇒ feature map size 감소
- 2×2 Max-pooling Layer (Stride 2) ⇒ 강하게 activate된 특징 선택
⇒ feature channels 2배 증가
- Bottle Neck
- 3×3 Convolution Layer + ReLu + Batch Normalization (No Padding, Stride 1) x 2
- Dropout Layer
- Upsampling
- 2×2 Transposed convolution layer (Stride 2)
→feature channel 1/2 감소
- Skip connection
: contracting path에서 동일한 level의 Feature Map을 추출하고 이전 Layer의 Feature Map을 Cropping한 후 Concatenate
padding 없는 convolution으로 인해 출력이미지는 입력보다 일정한 경계 너비만큼 작다. 따라서, 모든 convolution의 border pixels의 loss를 해결하기 위해 cropping이 필요하다.
→ context 정보를 담고 있는 decoder’s features에, encoding step에서 얻어서 decoding step의 맨처음 Input 크기에 맞춰 crop한 위치정보를 담은 encoder’s feature를 결합해 pixel perfect segmentation을 얻을 수 있음
- 3×3 Convolution Layer + ReLu + BatchNorm (No Padding, Stride 1) x 2
⇒ feature map size 감소
- last layer에서는 class의 개수만큼 필터를 갖고 있는 1x1 convolution을 사용해 각 픽셀이 어떤 class에 해당하는지 mapping해서 3차원벡터(w x h x class)를 생성한다.
⇒ Total network Layers: 23 convolutional layers( 18: 3x3conv + 4: 2x2up-conv + 1: 1x1conv )
⇒ 출력 segmentation map의 원활한 타일링을 허용하려면 모든 2x2 max pooling 작업이 ‘짝수’ x, y size의 레이어에 적용되도록 입력 타일 크기를 선택하는 것이 중요하다.
- Downsampling
Training
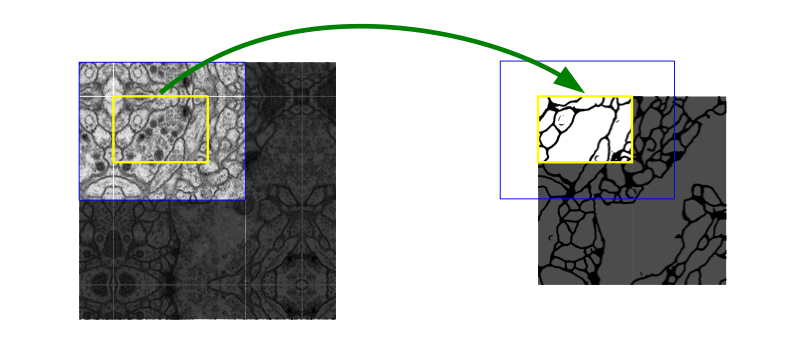
Overlap-tile strategy
본 논문의 U-Net 구조의 경우 패딩이 없기 때문에 매번 출력이미지가 더 작기 때문에 원하고자 하는 output보다 큰 input이 필요하다. 더불어 biomedical domain은 labeling이 어렵고 고해상도 이미지가 대부분이기 때문에 많은 연산량을 필요로 해 큰 이미지를 seamless segmentation할 수 있도록 overlap-tile 전략을 사용했다.
이미지를 Tile; Patch로 나눠서 입력으로 사용한다. → 파란 박스를 넣으면 노란 박스가 추출되는데 다음 Tile에 대한 Segmentation 을 얻기 위해 이전 입력의 일부분이 포함된다.
→ missing input data는 ‘mirroring’으로 보완된다.
Mirroring
이미지의 경계부분을 예측할 때 padding이 일반적이지만, 본 논문에서는 경계에 위치한 이미지를 복사하고 좌우반전 이미지를 생성한 후 주변에 붙여 input 값으로 사용했다.
Weight Loss _loss function
input image와 그에 대응되는 segmentation map은 Caffe 기반, SGD로 구현되었다.
overhead를 최소화하고 GPU 메모리를 최대한 활용하기 위해 큰 Batch size보다 큰 input tiles을 선호하므로, 배치를 single image로 줄이는 과정을 진행했다.
따라서 이전에 본 과거의 많은 수 훈련샘플이 현재 optimization 단계의 업데이트를 결정하도록 높은 momentum(0.99) 사용함
- 매 세포분할작업에서 challenge는 동일 클래스의 접촉하는 객체를 분리하는 것이기 때문에 접촉하는 세포 사이의 분리 배경 레이블에 loss function에 큰 weight를 부여하는 ‘weighted loss’를 제안했다.
- 즉, Loss func = pixel마다 구한 E func의 총합을 말한다.
softmax
$p_k(x)$: 픽셀 x가 클래스 k일 확률(0~1)
$a_k(x)$: 픽셀위치에서 feature channel ‘k’의 activation, 즉 픽셀 x가 class k일 값( K: 클래스의 수 )
즉, softmax는 activation $a_k(x)$가 최대인 k에 대해 거의 1에 가까워지고, 다른 모든 k에 대해서 거의 0에 가까워진다.
cross entropy
각 위치에서 E를 사용해 나온 편차를 페널티로 주는 역할을 한다.
$pl$의 $l$: 1부터 K까지의 각 픽셀의 실제 label, w: training 중 특정 픽셀 더 큰 중요도를 부여하기 위해 도입한 가중치 맵
각 ground truth segmentation에 대해 weight map을 미리 계산해서, training dataset에서 특정 클래스의 다른 픽셀 frequency를 보상하고, touching cells 사이에 작은 speration borders 를 네트워크가 학습하도록 하였다.
$w_c(x)$: 클래스 frequency를 균형맞추기위한 가중치맵
$d_1(x)$: 가장 가까운 세포의 경계까지의 거리
$d_2(x)$: 두번째로 가까운 세포의 경계까지의 거리
논문의 weight hyper parameter는 $w_0$=10, $\delta$=5 로 설정
→ 최종적으로, w(x)는 pixel x와 border의 거리가 가까우면 값이 커지므로, 해당 픽셀의 loss 비중이 커진다. 이 과정은 경계에 해당하는 pixel을 잘 학습하게 되서 accuracy 높인다.
⇒ 많은 convolution layers와 different paths가 있는 deep networks에서는 weights의 initialization가 매우 중요하다.
이 논문에서, 이상적인 initial weights는 네트워크의 각 feature map이 대략 단위 분산을 가지도록 조정했다. Gaussian distribution 정규분포인 $\sqrt{2/N}$ (N: 하나의 뉴런에 대한 입력 노드 수) 사용하여 3x3 convolution과 이전 레이어의 64개의 feature channels가 있다면 N은 9 x 64 = 576가 된다.
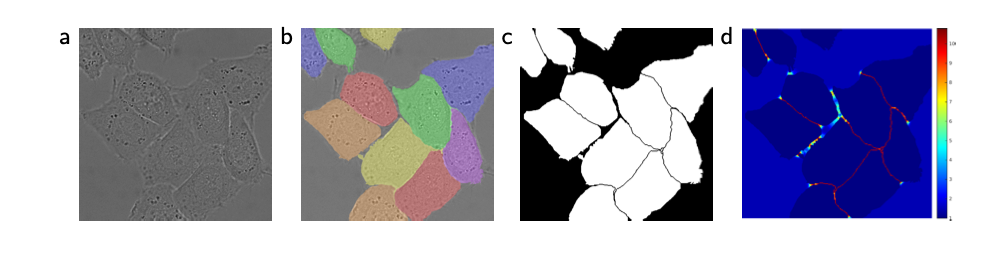 Cell Segmentation(DIC로 촬영한 HeLa 세포들) d. border pixels을 학습하도록 한 픽셀단위 loss weight map
Cell Segmentation(DIC로 촬영한 HeLa 세포들) d. border pixels을 학습하도록 한 픽셀단위 loss weight map
Data Augmentation
훈련 샘플이 적을 때, network invariance & robustness properties을 학습하는데 필수적인 과정이다.
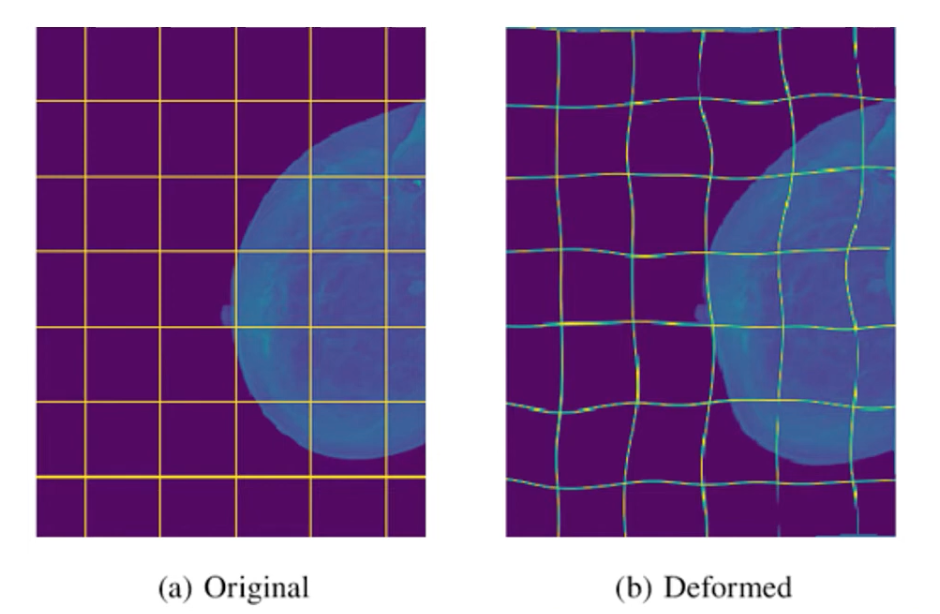
biomedical의 현미경 이미지의 경우, shift, rotation, gray value에 대한 robustness가 필요하다. 특히 훈련 샘플에 대한 random elastic deformation은 annotate된 이미지가 매우 적은 경우에 핵심개념이 되고 coarse 3x3 grid에서 무작위 변위 벡터를 사용해 smooth deformations을 생성한다.
Experiments
전자현미경 기록에서 신경구조 분할
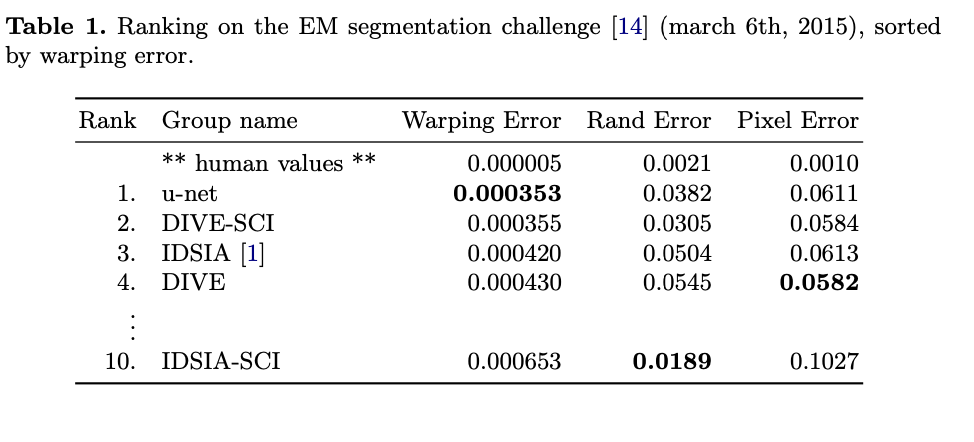
Dataset: ISBI 2012, EM segmentation challenge
training data: 초파리(Drosophila) 1령 유충의 복부 신경 코드(VNC)를 시리얼 섹션 투과 전자 현미경으로 촬영한 ‘30’장의 image(512x512 pixel) set
evaluation: map을 10개의 다른 레벨로 임계값을 설정한 후, Warping 오류’, ‘Rand 오류’, ‘Pixel 오류’를 계산했다.
U-Net(input data에 7가지 rotated version에 대해 평균화)은 추가적 전처리, 후처리없이 warping error: 0.0003529, rand error: 0.0382를 달성했다.
아래 IDSIA-SCI의 Rand Error는 sliding-window 기법인 3의 확률 맵에 dataset 특화된 pre-processing 방법을 적용한 경우이다.
광학 현미경 이미지에서의 세포 분할 작업
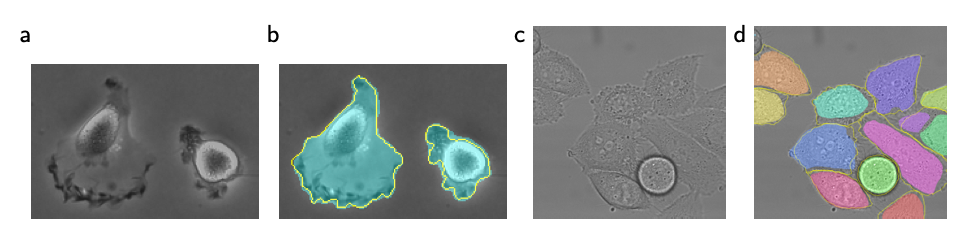
 a,b: ‘PhC-U373’ / c,d: ‘DIC-HeLa’
a,b: ‘PhC-U373’ / c,d: ‘DIC-HeLa’a,b 실험 과정
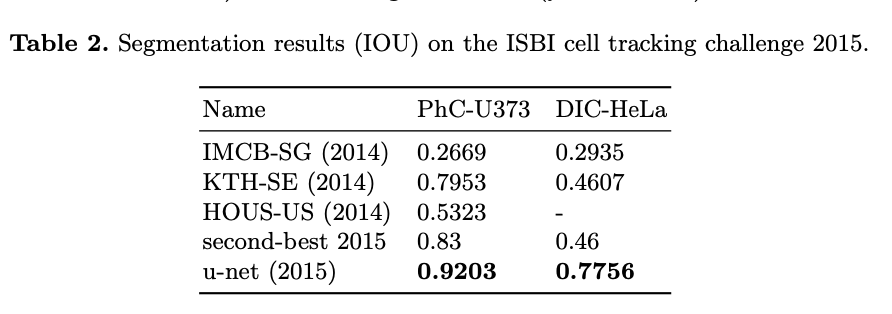
“PhC-U373”은 위상차 현미경으로 기록된 폴리아크릴아미드 기질 위의 U373 교모세포종-성상세포(Glioblastoma-astrocytoma) 세포를 포함하고 35개의 부분적 라벨링된 훈련이미지가 포함되었다.
⇒ 이 데이터 셋에서 평균 IOU 92%를 달성했다.(better than 83)
c,d 실험 과정
“DIC-HeLa”는 차등 간섭 대비(DIC) 현미경으로 기록된 평평한 유리 위의 HeLa 세포를 포함하고 20개의 부분적 라벨링된 훈련이미지가 포함되었다.
⇒ 이 데이터 셋에서 평균 IOU 77.5%를 달성했다.(better than 46)
Conclusion
- 다양한 biomedical segmentation 응용에서 매우 우수한 성능 달성했다. elastic deformation을 사용한 data augmentation 덕분에, annotated image가 매우 적게 필요하며, NVidia Titan GPU(6GB) 에서 training 시간이 단 10시간으로 매우 합리적인 결과를 보여줬다.